СБОР И ПОДГОТОВКА ДАННЫХ
Источник данных
Датасет содержит КТ-изображения легких, разделенные на пять классов:
- COVID
- Normal (здоровые легкие)
- Lung Opacity (помутнение легких)
- Viral Pneumonia (вирусная пневмония)
- Lung Cancer (рак легких)
Файлы изображений хранятся в папках, соответствующих этим классам.
Сам датасет содержит 27 188 пар изображений, которые включают сам КТ легких и маски с очагами заражения (рис. 1):
Рисунок 1. Легкие больного COVID-19
Преобразование данных
В коде реализована функция build_data_pipeline, которая выполняет несколько ключевых операций:
- Читает пути ко всем изображениям.
- Присваивает каждому изображению метку класса.
- Загружает изображения, выполняет их нормализацию и изменение размера (256x256 пикселей).
- Разделяет данные на обучающую (80%) и валидационную (20%) выборки.
- Оборачивает данные в
tf.data.Datasetи используетbatch()для группировки в пакеты по 4 изображения.
АРХИТЕКТУРА МОДЕЛИ
Для классификации изображений используется нейросеть, основанная на архитектуре VGG19. Основные этапы построения модели в функции build_model:
- Используется предобученная VGG19 с весами ImageNet, при этом исключается верхняя классификационная часть (
include_top=False). - Добавляется слой
GlobalAveragePooling2D(), который сворачивает выходные карты признаков в вектор. - Добавляется полносвязный слой из 256 нейронов с активацией ReLU.
- Применяется
Dropout(0.5), чтобы снизить переобучение. - Финальный выходной слой имеет 5 нейронов с функцией активации
softmaxдля предсказания вероятностей классов. - Модель компилируется с
Adam(learning rate = 1e-4) и функцией потерьsparse_categorical_crossentropy.
ОБУЧЕНИЕ МОДЕЛИ И ВЫЧИСЛИТЕЛЬНЫЕ РЕСУРСЫ
Функция train выполняет процесс обучения:
- Вызывает
build_data_pipeline()для загрузки данных. - Создаёт модель через
build_model(). - Обучает её в течение 150 эпох с проверкой на валидационном наборе.
- Использует
ModelCheckpointдля сохранения лучшей модели (поval_loss). - Добавляет
EarlyStoppingсpatience=10, чтобы прервать обучение при отсутствии улучшения.
Обучение продлилось в 21 эпоху из-за защиты от предобучения которое продемонстрировано на изображении 2:
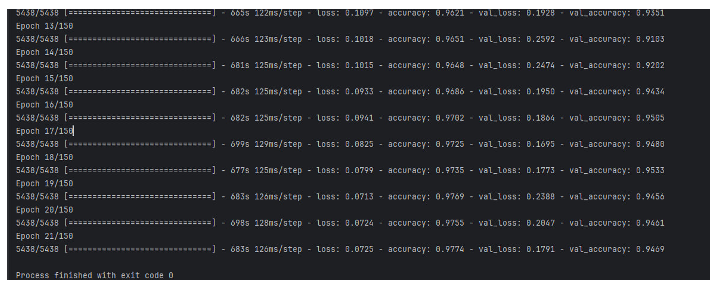
Рисунок 2. Количество пройденных эпох
ПРЕДСКАЗАНИЕ И ВИЗУАЛИЗАЦИЯ
Функция predict_and_show реализует предсказание для одного изображения:
- Загружает и предобрабатывает изображение аналогично обучающим данным.
- Делает предсказание через
model.predict(). - Выводит изображение с предсказанным классом и вероятностями всех классов в виде текста.
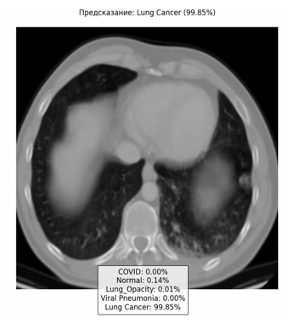
Рисунок 3. Предсказание рака легких
Выводы по обучению:
- Модель показывает высокую точность (~97% на обучении и ~94-95% на валидации).
- Функция потерь (Loss) постепенно уменьшается, что подтверждает улучшение модели.
- На 14-й и 20-й эпохах были скачки валидационного Loss, что может указывать на переобучение.
Но такой статистики для действительной оценки обученной модели недостаточно, требуется провести еще тесты. Поэтому для такой задачи был реализован программный код, который анализирует обученную модель, оценивает её по различным метрикам и проводит тестирование.
Функционал кода:
- Оценка модели на тестовом датасете (accuracy, loss)
- Подсчет метрик (Precision, Recall, F1-score)
- Матрица ошибок (confusion matrix)
- ROC-кривая и AUC
- Гистограмма предсказаний
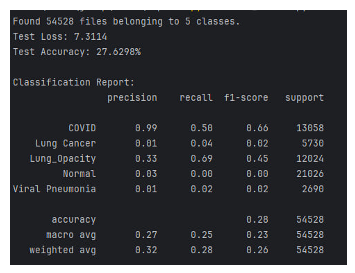
Рисунок 4. Оценка модели и f1-score
1. Низкая общая точность (27.63%)
Модель плохо справляется с классификацией, особенно для классов "Lung Cancer", "Normal" и "Viral Pneumonia".
2. Дисбаланс классов
Класс "Normal" имеет 21,026 примеров, а "Viral Pneumonia" — всего 2,690. Это приводит к смещению модели в сторону доминирующих классов.
3. Плохие метрики для отдельных классов
- Lung Cancer: Precision = 0.01, Recall = 0.04
- Viral Pneumonia: Precision = 0.01, Recall = 0.02
Модель почти не различает эти классы.
Classification Report:
Precision (Точность):
- COVID: 0.99 — почти все предсказанные COVID-случаи верны.
- Lung Cancer: 0.01 — 99% предсказаний рака ошибочны.
- Viral Pneumonia: 0.01 — модель почти всегда ошибается.
Recall (Полнота):
- COVID: 0.50 — модель пропускает 50% реальных COVID-случаев.
- Lung Cancer: 0.04 — обнаруживает только 4% реальных случаев.
- Normal: 0.00 — полностью игнорирует здоровые легкие.
F1-score:
- Максимальное значение у COVID (0.66) — лучший результат.
- Остальные классы близки к нулю.
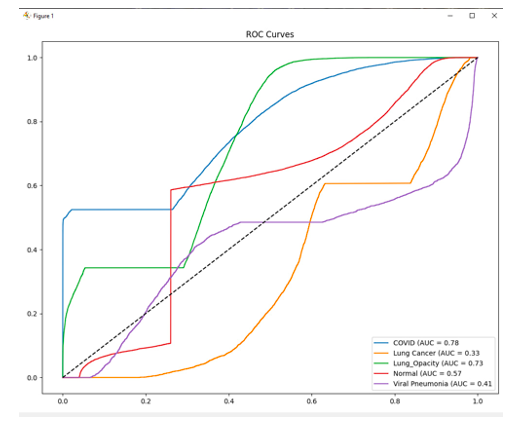
Рисунок 5. ROC Curves
ROC-кривые и AUC:
- COVID (AUC = 0.78):
Качество разделения приемлемое, но далеко от идеала (AUC > 0.9). Кривая значительно выше диагонали. - Lung Cancer (AUC = 0.33):
Кривая близка к случайному угадыванию (AUC ≈ 0.5). Модель не отличает рак от других заболеваний. - Lung_Opacity (AUC = 0.73):
Среднее качество. Модель частично выделяет случаи помутнения, но часто ошибается. - Normal (AUC = 0.57):
Модель не различает здоровые легкие. - Viral Pneumonia (AUC = 0.41):
Качество хуже случайного (AUC < 0.5). Модель путает пневмонию с другими классами.
Интерпретация AUC:
- AUC = 1: Идеальное разделение классов.
- AUC = 0.5: Случайное угадывание.
- AUC < 0.5: Модель работает хуже случайного угадывания (проблема в обучении).
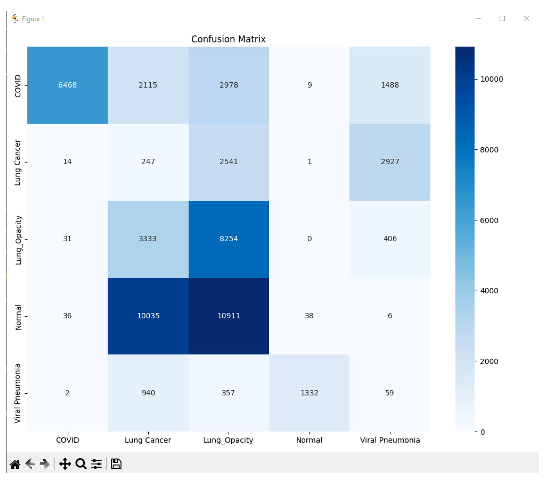
Рисунок 6. Матрица ошибок
Матрица ошибок (Confusion Matrix):
- COVID:
- Истинно положительные (TP): 6468 (модель правильно определила COVID в 6468 случаях).
- Ложные отрицательные (FN): 2115 (модель пропустила COVID в 2115 случаях).
- Ложные положительные (FP): 2978 (модель ошибочно присвоила класс COVID другим заболеваниям в 2978 случаях).
- Lung Cancer:
- TP: 14 (крайне мало верных предсказаний).
- FN: 247 (много пропущенных случаев рака).
- FP: 2541 (модель часто путает рак с другими классами).
- Normal:
- TP: 31 (почти не распознает здоровые легкие).
- FN: 3333 (пропускает большинство здоровых случаев).
- FP: 8254 (часто ошибочно помечает другие классы как "Normal").
- Viral Pneumonia:
- TP: 36 (очень низкое качество распознавания).
- FN: 10035 (модель не видит пневмонию).
- FP: 10911 (постоянные ложные срабатывания).
Итог:
- Модель переобучена на класс COVID (высокий TP, но много FP).
- Не распознает Lung Cancer и Viral Pneumonia (TP близки к нулю).
- Для классов Normal и Lung_Opacity наблюдается сильный дисбаланс (много FP/FN).
Рекомендации к улучшению:
1. Балансировка данных: использовать взвешенные лосс-функции для учета дисбаланса:
class_weights = compute_class_weight('balanced', classes=np.unique(y_train), y=y_train)
model.fit(..., class_weight=class_weights)
Применить аугментацию для малых классов (повороты, отражения, изменение контраста).
2. Оптимизация архитектуры модели:
- Возможно стоит заменить VGG19 на EfficientNetB4 — у нее есть современная архитектура, лучше работающая с медицинскими изображениями.
- Добавить слои Dropout (0.5) и Batch Normalization для борьбы с переобучением.
3. Настройка гиперпараметров
- Использовать оптимизатор AdamW с learning rate 3e-5.
- Добавить L2-регуляризацию в плотные слои.
Использование ресурсов
На системе с характеристиками:
- ОЗУ: 32 ГБ DDR4
- Процессор: AMD Ryzen 5 3600X
- Видеокарта: GTX 1660 Super (6 ГБ VRAM)
Во время загрузки изображений потребляется до 16 ГБ оперативной памяти. Во время самого обучения используется 3-4 ГБ ОЗУ и до 5 ГБ видеопамяти.
Обучение модели требует значительных вычислительных ресурсов. Для повышения качества классификации можно увеличить размер изображений, например, до 512x512 пикселей, однако это приведёт к увеличению потребления памяти. Для ускорения обучения можно повысить BATCH_SIZE, но это также потребует более мощного оборудования, включая видеокарту с большим объемом памяти и высокопроизводительный процессор.
ВЫВОД
Результаты экспериментов показывают, что применение глубокого обучения в диагностике заболеваний лёгких на КТ-изображениях позволяет автоматизировать процесс выявления патологий. Хотя, несмотря на высокую точность модели на обучающем датасете, её реальная эффективность на тестовых данных оказалась ниже ожидаемой. Анализ метрик F1-score, confusion matrix и ROC-кривых выявил проблемы с классификацией отдельных заболеваний, особенно рака лёгких и вирусной пневмонии.
Для улучшения результатов необходимо:
- Оптимизировать баланс классов в датасете, так как текущая модель хуже распознаёт малочисленные категории.
- Рассмотреть альтернативные архитектуры нейросетей, такие как ResNet, EfficientNet или Vision Transformers, которые могут дать более стабильные предсказания.
- Применить методы увеличения данных (Data Augmentation) и более сложные параметры регуляризации для повышения качества предсказания модели.
- Провести дополнительные тесты на независимых медицинских датасетах, чтобы оценить адаптивность модели к реальным условиям.
Таким образом, исследуемая модель демонстрирует потенциал в автоматизированной диагностике, но требует доработки и более глубокой оценки перед клиническим применением.
Список литературы
- Симонян, К., Зиссерман, А. Очень глубокие сверточные сети для распознавания изображений [Текст] / К. Симонян, А. Зиссерман. // arXiv preprint arXiv:1409.1556, 2014
- Кингма, Д.П., Ба, Дж. Adam: Метод стохастической оптимизации [Текст] / Д.П. Кингма, Дж. Ба. // International Conference on Learning Representations (ICLR), 2015
- Чоудхури, М., Рахман, Т. База данных COVID-19 Radiography Database [Текст] / М. Чоудхури, Т. Рахман и др. // Kaggle, 2020. – URL: https://www.kaggle.com/tawsifurrahman/covid19-radiography-database



